
Profil ADN SNP (ISAG 2020) + Pedigree Chien
Commandez l'examen
Description
Avec cette nouvelle norme, plus de 200 marqueurs SNP sont mis en evidence (contre 22 seulement avec l’ancienne norme ISAG 2006), ce qui permet non seulement de confirmer la parenté avec une plus grande certitude, mais aussi de calculer l’hétérozygotie (diversité génétique) et un coefficient de consanguinité (degré de parenté). Deux outils importants pour maintenir la santé génétique d’une population.
Attention: la norme ISAG 2020 basée sur les SNP n’est pas compatible avec l’ancienne norme ISAG 2006 basée sur les STR. Tous les animaux impliqués dans un contrôle de parenté doivent avoir le même type.
Espèce cible et races
Chien
Toutes les races
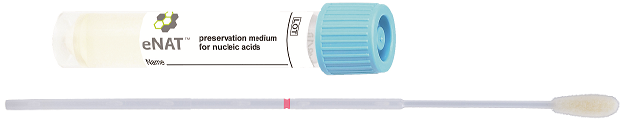
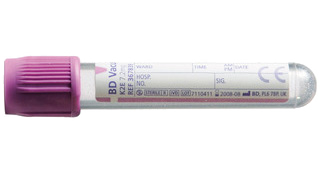
Prélèvement
- Ecouvillon génétique
- EDTA sang complet
Délai
14 jours
