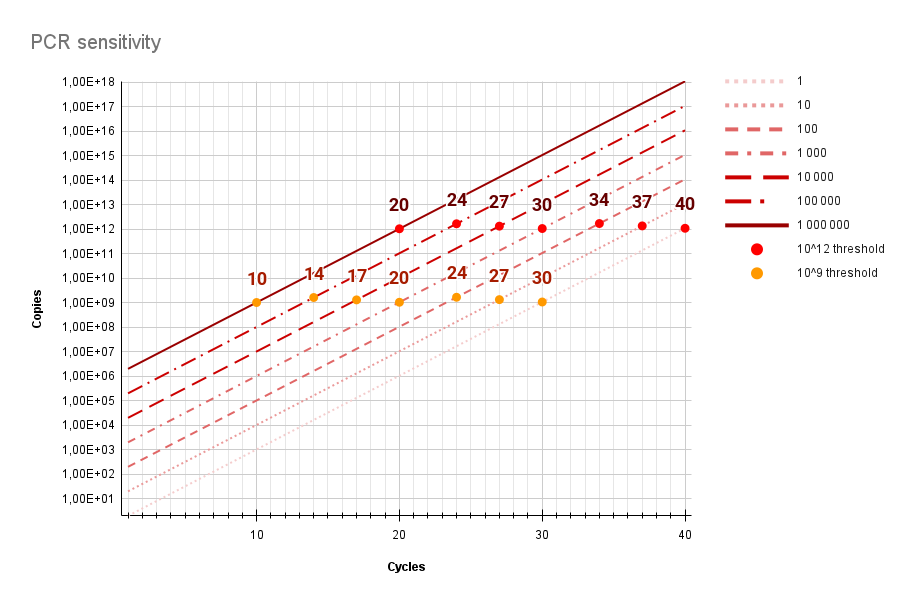
Théoriquement, après 40 cycles, 1 morceau de code génétique était présent au départ de l’analyse a produit 240 ou environ mille milliards (1.099.511.627.776 pour être exact) copies.
Dans le graphique ci-dessous montre l’évolution du cycle (Ct) où il y avait initialement 1, 10, 100, 1.000, 10.000, 100.000 et 1.000.000 particules présentes dans le’échantillon, et ceci quand on met le seuil (threshold) à 1012copies (rouge) ou à 109 copies (orange).
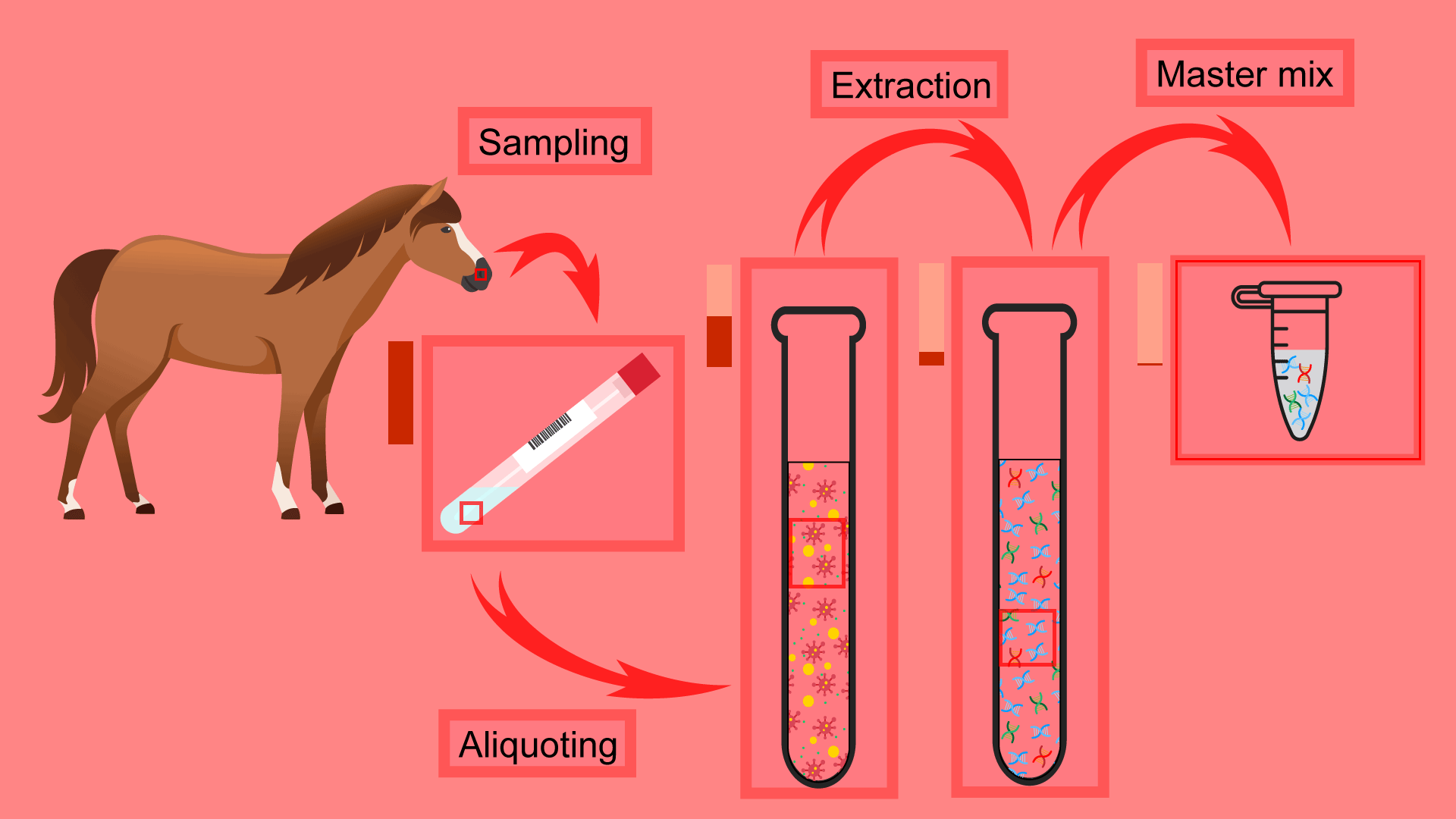
Notez bien que le patient n’est pas analysé en entier, mais qu’un échantillon prélevé, transporté et conservé dans les meilleures conditions en attendant l’analyse.
Tout l’échantillon n’est pas analysé, mais seulement une toute petite fraction. De cette fraction, le matériel génétique est extrait dans son intégralité. Ce pré-traitement n’est pas 100% efficace, une partie est toujours perdue.
Enfin, seule une fraction du matériel génétique extrait est ajoutée aux réactifs (le master mix) pour initialiser la réaction.
Le graphique présume une efficacité de réaction de 100%, chaque cycle conduit exactement à un doublement. En réalité, c’est un peu moins.
En d’autres termes, au cours des manipulations du patient au tube à essai beaucoup de matériel est perdu ou non utilisé. La sensibilité pour détecter 1 à 10 particules n’est pas excessive car 1 particule dans le tube à essai peut signifier un multiple chez le patient.
La question: “Y a-t-il un lien entre la valeur CT et le nombre de particules infectieuses présentes chez le patient?” elle n’est donc pas si simple à répondre. En théorie, on pouvait compter en arrière, mais le résultat dépend fortement de la qualité de l’échantillon, de l’efficacité de l’extraction du matériel génétique et de l’efficacité de la réaction PCR. Aucun de ceux-ci n’est à 100%.
