Non, les deux profils sont basés sur des technologies complètement différentes.
Pour la vérification de la filiation, les profils ISAG 2006 ne peuvent être comparés qu’avec d’autres profils ISAG 2006 et les profils ISAG 2020 ne peuvent être comparés qu’avec des profils ISAG 2020.
En outre, pour la vérification de la filiation, Zoolyx, VHL et Certagen acceptent certains profils ISAG 2020 émis par l’un de ces trois laboratoires.
| Chiot | Parents | Vérification du pedigree |
| ISAG 2006 | ISAG 2006 (Tout labo) | Possible |
| ISAG 2006 | ISAG 2020 (Tout labo) | Impossible, commandez pour le chiot H3209 ISAG 2020 |
| ISAG 2020 (Zoolyx/VHL/Certagen) | ISAG 2020 (Zoolyx/VHL/Certagen) | Possible |
| ISAG 2020 (Zoolyx/VHL/Certagen) | ISAG 2020 (Autre labo) | Impossible, commandez pour les parents H3200 ISAG 2020 |
| ISAG 2020 (Zoolyx/VHL/Certagen) | ISAG 2006 (Tout labo) | Impossible, commandez pour les parents H3200 ISAG 2020 |
| Pas de profil | ISAG 2006 (Tout labo) | Commandez pour le chiot H3201 ISAG 2006 ou H3204 ISAG 2006+2020 si futur animal reproducteur |
| Pas de profil | ISAG 2020 (Zoolyx/VHL/Certagen) | Commandez pour le chiot H3209 ISAG 2020 |
| Pas de profil | ISAG 2020 (Autre labo) | Commandez pour le chiot H3209 ISAG 2020 et pour les parents H3200 ISAG 2020 |
| Pas de profil | Pas de profil | Commandez pour le chiot H3209 ISAG 2020 et pour les parents H3200 ISAG 2020 |
ISAG 2006 STR
Cette série utilise 21 marqueurs STR (Short Tandem Repeats), de courts morceaux d’ADN qui se répètent et dont la longueur totale peut varier d’un individu à l’autre. Limitations
- Informations génétiques limitées: Avec seulement 21 marqueurs, l’application est limitée à l’établissement de la paternité/maternité/parentalité.
- Filiations « problématiques »: Dans certains cas, un profil ADN de la mère est néanmoins nécessaire pour confirmer la paternité.
ISAG 2020 SNP
Cette série utilise 230 marqueurs SNP (Single Nucleotide Polymorphism), des nucléotides uniques situés à des endroits bien définis du génome et répartis sur l’ensemble des chromosomes, qui peuvent différer d’un individu à l’autre. Avantages
- Résolution plus élevée: étant donné le plus grand nombre de marqueurs, les SNP offrent un profil génétique plus complet.
- Plus stable: les SNP souffrent généralement moins de mutations spontanées que les STR et donnent donc des résultats plus cohérents.
- Application plus large: les SNP peuvent être utilisés en plus de la filiation pour l’identification de la race, la vérification de la fratrie et le calcul de l’hétérozygotie et du coefficient de consanguinité.
Pour chaque fichier, des frais de gestion sont également ajoutés à la facture. Il s’agit de tous les coûts qui ne sont pas directement liés aux analyses. Il s’agit du traitement administratif, du ramassage, de la logistique et des frais d’expédition, …
Ce frais est facturé au maximum une fois par jour et par propriétaire. Si nous recevons plusieurs échantillons du même propriétaire en même temps, ce coût ne sera facturé qu’une seule fois.
Sur la page de votre rapport, vous pourrez également voir le prix de ces enquêtes. Ceci est juste pour votre information. Il n’est pas possible de régler ce solde en ligne. Une fois tous les examens terminés, la facture est préparée et envoyée par la poste.
Sur la facture que vous avez reçue de Zoolyx, vous trouverez tous les détails pour effectuer le paiement. Dans la case en haut à gauche, vous trouverez notre IBAN, le BIC de notre banque et notre numéro de TVA.
IBAN: BE 69 3631 3257 3778
BIC: BBRUBEBB
TVA: BE0549.932.689
Pour que votre paiement soit enregistré correctement, vous devez également joindre un message structuré à votre paiement. Vous le trouverez dans la case grise avec le montant à payer.
Zoolyx est un laboratoire de diagnostic vétérinaire. Les vétérinaires et les cabinets vétérinaires nous demandent de collecter et d’analyser des échantillons pour les aider dans le diagnostic et le suivi de leurs patients. Si vous avez reçu une facture de notre part, c’est probablement parce que nous avons reçu vos coordonnées ainsi qu’un échantillon de votre vétérinaire.
Pour toute question relative aux résultats ou à leur interprétation, veuillez contacter votre vétérinaire.
Si vous avez reçu une facture contenant des données incorrectes ou manquantes, veuillez en informer facturation@zoolyx.be dans les deux mois suivant la date de la facture originale et vous recevrez une version corrigée..
Vous avez reçu une facture parce que nous avons effectué une analyse à la demande de votre vétérinaire. Bien entendu, vous aurez également accès aux résultats.
Si nous avons également reçu votre adresse électronique du vétérinaire, vous pouvez vous connecter à notre site web zoolyx.be pour accéder à vos résultats.
Si nous n’avons pas reçu votre adresse électronique, vous pouvez demander au vétérinaire demandeur de vous communiquer les résultats.
Lié au sexe – Autosomique
Si un gène est situé sur un chromosome sexuel (X ou Y), on parle de trait lié au sexe. S’il est situé sur l’un des nombreux autres chromosomes, on parle de trait autosomique. La grande majorité des traits sont autosomiques. Les traits liés au sexe chez les mammifères sont principalement liés au chromosome X, seuls quelques-uns sont liés au chromosome Y. Un trait autosomique est exprimé de manière égale dans les deux sexes. Un trait lié au chromosome X se comporte chez les individus de sexe féminin comme un trait autosomique, puisqu’elles possèdent deux chromosomes X. Dans ce cas, une variante peut être partiellement ou totalement compensée par une autre variante sur l’autre chromosome X. Les individus de sexe masculin, en revanche, n’ont qu’un seul chromosome X. Une variante ou un défaut s’y exprimera toujours. Par ailleurs, un trait lié au chromosome Y ne peut s’exprimer que chez les individus mâles, les femelles n’ayant pas de chromosome Y. Ce dernier point est quelque peu théorique: aucun trait lié à l’Y ayant une utilité pratique (c’est-à-dire pouvant être testé) n’est actuellement connu chez nos animaux de compagnie.
Dominant – Co-dominant – Récessif
Une deuxième distinction reflète la mesure dans laquelle une caractéristique est exprimée. Il suffit que la variante soit présente sur l’un ou l’autre gène pour qu’elle soit dominante. Si la variante ne s’exprime que lorsque les deux gènes sont porteurs de la variante, celle-ci est récessive. Une troisième possibilité est que la variante ne soit que partiellement exprimée. Il existe alors trois formes: celle où le trait est pleinement exprimé parce que les deux gènes portent la variante, celle où il n’est pas exprimé simplement parce que la variante n’est pas présente, et celle qui se situe entre les deux, où seul l’un des gènes présente la variante. Dans ce cas, on parle de codominance ou de dominance partielle. Les traits liés à l’X (et à l’Y) sont toujours dominants chez les individus de sexe masculin.
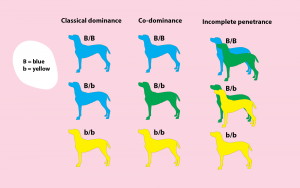
Pénétrance
La présence d’un variant ne garantit pas toujours son expression phénotypique, même lorsqu’il est dominant ou, dans le cas de la récessivité, présent sur les deux gènes. Dans ce cas, on parle de pénétrance incomplète d’une variante. Cela ne se produit pas pour tous les défauts ou traits génétiques, mais peut expliquer pourquoi la génétique n’est pas toujours une mathématique exacte, bien qu’elle soit parfois présentée comme telle. Plusieurs mécanismes expliquent ce phénomène:
- plusieurs gènes (polygéniques) déterminent le phénotype, un gène pouvant influencer, voire annuler l’effet d’un autre;
- le génotype ne s’exprime lorsque certains facteurs environnementaux (mode de vie, alimentation, stress…) interviennent et/ou à un certain âge;
- le génotype est activé ou désactivé en fonction des conditions (régulation épigénétique) vécues par l’individu lui-même ou ses ancêtres. Ainsi, une préférence préprogrammée pour l’activation de l’allèle paternel ou maternel, ou vice versa, peut également apparaître (empreinte génomique).
La biobliothèque génétique de chaque individu est organisé en deux séries de chromosomes: l’une provenant du père, l’autre de la mère. Tous les chromosomes ont un homologue dans l’autre série. Par homologue, on entend qu’il s’agit d’une copie fonctionnelle, c’est-à-dire qu’ils possèdent les mêmes gènes (à l’exception du chromosome Y), mais pas d’une copie exacte. Au sein d’une même espèce, il existe souvent deux variantes ou plus d’un même gène. Par exemple, la variante du gène sur le chromosome du père peut être différente de la variante du gène de la mère. Une telle variante est également appelée allèle. Chaque individu possède donc deux allèles pour chaque gène. Ceux-ci peuvent être identiques, homozygotes, ou différents, hétérozygotes. Le génotype est une description (codée) des variantes présentes dans les deux gènes. Par exemple, A/A ou a/a ou A/a.
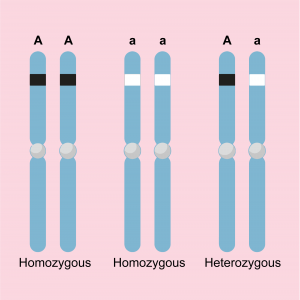
Représentation schématique de deux chromosomes homologues contenant un gène avec deux allèles possibles et donc trois génotypes possibles : A/A, a/a (tous deux homozygotes) et A/a (hétérozygote).
Le phénotype, quant à lui, est une description (verbale) du trait tel qu’il est observé chez l’individu; par exemple, noir, brun, jaune… Plusieurs génotypes peuvent conduire au même phénotype et vice versa. Le phénotype peut souvent être déduit du génotype; inversement, on a parfois besoin du phénotype pour déterminer le génotype avec plus de précision. C’est par exemple le cas lorsqu’un phénotype est contrôlé par plusieurs gènes, mais que ceux-ci ne sont pas tous connus au niveau de l’ADN et ne peuvent donc pas être testés.
Un exemple permet d’y voir plus clair
Les trois couleurs principales du pelage des labradors sont le jaune, le brun (chocolat) et le noir (il en existe d’autres, mais pour ne pas trop compliquer les choses, nous nous arrêterons là). Le phénotype de la couleur de la robe est contrôlé par deux gènes ou loci (singulier: locus) à transmission dominante/récessive classique : le locus B et le locus E. Ces deux loci ont chacun deux allèles possibles, respectivement B soit b, et E soit e. Le locus B détermine si le pigment formé est noir (B) ou brun (b), tandis que le locus E active (E) ou désactive (e) le locus B. Il en résulte 9 génotypes possibles:
 |
E/B | BB | Bb | bb |
| EE | BBEE | BbEE | bbEE | |
| Ee | BBEe | BbEe | bbEe | |
| ee | BBee | Bbee | bbee |
Le tableau permet de déduire que tous les Labradors jaunes sont « fixés » pour le locus E avec l’allèle e. Dans ce cas, vous pouvez donc déduire le génotype du locus E (ee) du phénotype (jaune).
Si vous soumettez un prélèvement pour analyse en utilisant votre adresse e-mail, un compte sera créé automatiquement. Vous recevrez un message par e-mail pour activer le compte et mettre un mot de passe. Si vous faites cela en tant que vétérinaire, vous aurez un accès immédiat à toutes les fonctions vétérinaires.
Vous pouvez également vous inscrire en utilisant le menu myZoolyx > Se connecter > Enregistrer. Ensuite suivez ces étapes.
- Entrez votre adresse email.
- Attendez l’e-mail d’activation (regardez aussi dans spam s’il attend depuis plus d’une minute)
- Cliquez sur le lien ou le bouton d’activation dans le mail.
- Mettez un mot de passe. Lorsque vous tapez, le niveau de sécurité du mot de passe est indiqué. Tant que cela ne suffit pas, vous ne pouvez pas l’enrégistrer.
- Répétez le mot de passe et enregistrez.
- Connectez-vous et completez vos coordonnées.
- (vétérinaire seulement) Entrez votre numéro NGROD (nl) ou CRFOMV (fr).
- (vétérinaire seulement) Lorsque nous aurons vérifié votre enrégistrement en tant que vétérinaire, vous recevrez un email de confirmation. A partire de là, vous pouvez accéder à toutes les fonctions vétérinaires.
Vous pouvez suivre le colis via track.bpost.be. Entrez sur ce site le code dans l’e-mail que vous avez reçu.
Si la recherche ne produit pas de résultat ou si le colis reste dans un certain statut pendant plus de 5 jours, veuillez nous contacter en indiquant le numéro de dossier ou le code dans l’e-mail..
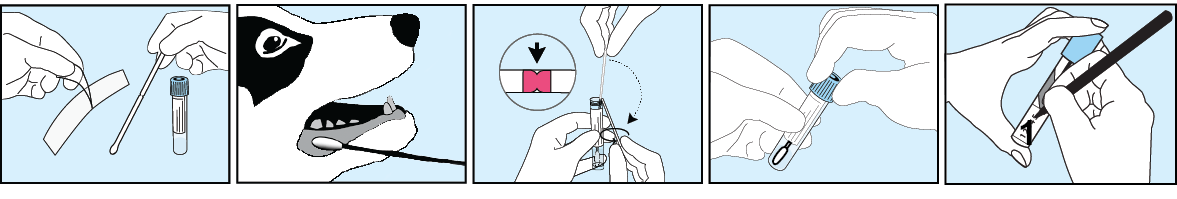
Pour les meilleurs résultats il est recommandé de ne pas nourrir ni abreuver les animaux 30 minutes avant le prélèvement.
- Prennez un animal à la fois.
- Faites tourner l’écouvillon pendant 30 sec sur une place qui se remplit de salive de façon naturelle: la muqueuse de la joue ou sous la langue. Empêchez que l’animal ne morde ou mâche l’éponge.
- Dévissez le bouchon du tube sans renverser le liquide, et mettez le baton dans la tube sans toucher l’extrémité. Puis brisez-le au niveau du point de cassure en pliant puis tournant le baton.
- Jetez la pièce casée. Revissez le bouchon fermement sur le tube, ne pas agiter.
- Collez une étiquette codebar de la micropuce sur le formulaire et marquez le numéro de série ou micropuce sur le tube. Si plusieurs nichées sont prélevées en même temps, marquez égallement la nichée.
- Demandez un assistant de tenir l’animal. La procédure n’est pas douloureuse pour l’animal, mais les animaux anxieux peuvent toujours être effrayés.
- Tournez un doigt autour des quelques poils de la crinière et tirez-le rapidement et puissamment.
- Les racines des poils sont visibles sous forme de points blancs à la base de la tige. Ne touchez pas.
- Mettre les poils dans un sachet plastique avec fermeture zip et sceller.
- Mettez un identifiant sur le sachet.
![]()
Pour les meilleurs résultats il est recommandé de ne pas nourrir ni abreuver les animaux 30 minutes avant le prélèvement.
- Désemballez l’écouvillon. Laissez la partie éponge bien attachée au tube.
- Faites tourner l’écouvillon pendant 30 sec sur une place qui se remplit de salive de façon naturelle: la muqueuse de la joue ou sous la langue. Empêchez que l’animal ne morde ou mâche l’éponge.
- Dévissez l’éponge du tube sans renverser le liquide, et vissez la inversement de sorte que l’éponge repose dans le liquide. Serrez bien.
- Retournez le tube 10x, ne pas agiter.
La Société Internationale de Génétique Animale (ISAG) définit la méthode et l’ensemble des marqueurs génétiques utilisés au niveau international pour vérifier la filiation des animaux. Pour les chiens, deux systèmes sont actuellement en place: ISAG 2006 et ISAG 2020. Ils ne sont PAS interchangeables. Le système ISAG 2006 sera progressivement abandonné.
ISAG 2006
Cette série utilise 21 marqueurs STR (Short Tandem Repeats), de courts morceaux d’ADN qui se répètent et dont la longueur totale peut varier d’un individu à l’autre.
|
||||||||||||||||||||||
| (*) le marqueur de sexe (**) a été supprimé fin 2017 et n’est plus pris en compte. |
Un profil ISAG 2006 valide
- est facile à lire, tous les chiffres doivent être lisibles sans ambiguïté;
- décrit les allèles des marqueurs sous la forme d’un nombre (par exemple 220/224). Le marqueur AMELOGENIN peut être indiqué comme X/X ou X/Y (ou Y/X);
- contient 22 (y compris FH2054) ou 21 marqueurs (à l’exclusion de FH2054) se compose d’au moins 20 marqueurs avec résultat (à l’exclusion de FH2054).
ISAG 2020
Cette série utilise 230 marqueurs SNP (Single Nucleotide Polymorphism), des nucléotides uniques situés à des endroits bien définis du génome et répartis sur tous les chromosomes, qui peuvent différer d’un individu à l’autre. L’ensemble se compose de deux panels, un panel standard dont les marqueurs sont présentés dans le tableau ci-dessous et un panel d’extension (non illustré) utilisé pour la filiation lorsque le panel standard ne fournit pas une certitude suffisante et pour le calcul de la variation génétique et des coefficients de consanguinité. Le résultat de chaque marqueur est le nucléotide (A, T, C ou G) présent sur un chromosome et sur l’autre. Par exemple A/G.
| AH0JFQR | BICF2G630111735 | BICF2P1159837 | BICF2P65087 | BICF2S23648905 |
| AH0JFQS | BICF2G630122583 | BICF2P1192522 | BICF2P728698 | BICF2S23713161 |
| AH1SDW1 | BICF2G630133028 | BICF2P1216677 | BICF2P774003 | BICF2S23737033 |
| AH1SDWZ | BICF2G630149030 | BICF2P1226745 | BICF2P805553 | G1425f16S28 |
| AH3999C | BICF2G630159183 | BICF2P1308802 | BICF2P878175 | P14 |
| AH704R2 | BICF2G630200354 | BICF2P1344095 | BICF2P885380 | P24 |
| AH892YB | BICF2G630209373 | BICF2P1346673 | BICF2P935470 | P27 |
| AHBKGAB | BICF2G630220326 | BICF2P1357746 | BICF2P990814 | P30 |
| AHBKGAE | BICF2G630271966 | BICF2P1454500 | BICF2P998036 | P37 |
| AHCTEGK | BICF2G630276039 | BICF2P157421 | BICF2S22943825 | P41 |
| AHD2CMU | BICF2G630326688 | BICF2P182473 | BICF2S23018785 | P54 |
| AHFBAS1 | BICF2G630409193 | BICF2P237994 | BICF2S23049416 | P55 |
| AHGJ8ZA | BICF2G630437783 | BICF2P251850 | BICF2S23111132 | P65 |
| AHHS65F | BICF2G630552597 | BICF2P277987 | BICF2S23124313 | P73 |
| AHKA3HV | BICF2G630558437 | BICF2P347679 | BICF2S23126079 | P80 |
| AHMSZUB | BICF2G630594648 | BICF2P382742 | BICF2S23214514 | P87 |
| AHPAV6S | BICF2G630634836 | BICF2P516667 | BICF2S23246455 | P88 |
| AHPAV6U | BICF2G630653298 | BICF2P554817 | BICF2S23326150 | TIGRP2P316532_rs8597522 |
| AHRSSI9 | BICF2G630689403 | BICF2P561057 | BICF2S23329382 | TIGRP2P356245_rs8830240 |
| AHUAOVN | BICF2G630708384 | BICF2P600196 | BICF2S2338108 | TIGRP2P362535_rs9130694 |
| AHVJM1W | BICF2G630798972 | BICF2P615597 | BICF2S23449478 | TIGRP2P372104_rs9153277 |
| Amelogenin | BICF2P1010945 | BICF2P624936 | BICF2S23535154 | TIGRP2P407751_rs8803124 |
| BICF2G630102146 | BICF2P105070 | BICF2P635478 | BICF2S236196 |
2 semaines, si un profil ISAG valide est disponible des deux parents.
La procédure peut prendre plus de temps si
- un profil est absent d’un ou des deux parents;
action: vous recevrez un email avec la demande de transmettre un profil ISAG valide. - le prélèvement d’un ou de plusieurs animaux est inadéquat;
action: vous recevrez un nouveau kit de prélèvement accompagné d’une demande de reprélever les animaux en question. Si le nouveau prélèvement s’avère également inadéquat, un échantillon de sang EDTA sera demandé. Aucun kit n’est envoyé pour cela; tout ce qu’il faut fait partie de l’équipement standard de votre vétérinaire.
Si un animal parent est décédé avant qu’un profil ADN ISAG compatible soit établi, il reste deux possibilités.
Il y a encore du matériel disponible.
Le profil ADN peut être établi sur la base de sperme (congelé) ou d’une biopsie.
Des brosses, jouets, colliers et autres objets personnelles, ainsi que du sang ou d’urine NE SONT PAS appropriés. La méthode d’analyse est optimisée pour les types de prélèvement purs. Des prélèvements contaminés nécessitent des techniques médico-légale qui sont beaucoup plus coûteuses sans aucune garantie de résultat.
Aucun échantillon n’est disponible.
Si le corps (ou le sperme) n’est plus disponible, le profil peut être reconstruit sur la base de:
- le profil des DEUX parents de l’animal décédé
- et plusieurs chiots dont l’animal décédé est considéré comme parent
Un profil reconstruit n’est pas toujours complet et contient des incertitudes. Plus de descendants (chiots) impliqués dans la reconstruction, plus le profil est correct et plus sûr la descendance.
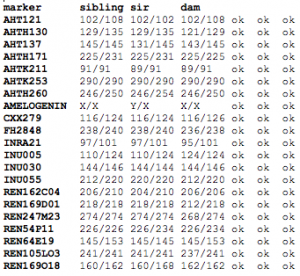
Le pedigree est accepté et rapporté comme étant JJJ si les situations suivantes apparaissent dans les 21 marqueurs examinés de la progéniture:
- un pedigree concluant: de tous les marqueurs de la progéniture un allèle se retrouve chez le père et l’autre chez la mère.
 p.ex. pedigree concluant
p.ex. pedigree concluant - un décalage est accepté: un allèle d’un marqueur ne se retrouve pas chez un des parents.
 p.ex. un décalage par rapport au père
p.ex. un décalage par rapport au père
Lorsque deux ou plusieurs marqueurs échouent, le pedigree est rejeté et rapporté comme
- JN = le père ne peut être exclu, la mère est exclue
 p.ex. 4 marqueurs échouent par rapport à la mère
p.ex. 4 marqueurs échouent par rapport à la mère - NJ = le père est exclu , la mère ne peut être exclue
- NN = père et mère sont exclus
Dans le cas aucun autre parent n’est éligible, le profil de l’animal parent présumé peut être rétablis par un nouveau prélèvement pour exclure des erreurs dans son profil initial.
Important: le pedigree se fait pour toute la nichée. Autant qu’un des chiots ne se qualifie pas, le pedigree des autres chiots, même qualifiés, ne sera pas validé.
Pour prouver la descendance, certaines règles doivent être respectées.
Les causes les plus fréquentes d’un rejet (provisoire) sont les suivantes:
- vraiment un autre père ou un double accouplement (certaines races sont notoires pour cela)
- une erreur dans deux ou plusieurs marqueurs du profil d’un ou des deux parents
- des mauvais parent(s) spécifié lors de la déclaration de la nichée
- le profil d’un ou des deux parents n’est pas ISAG compatible
- en cas de nichées simultanées, les chiots/chatons qui ont rampé dans une autre nichée
- en cas de prélèvement de nichées multiples en même temps, confusion des chiots/chatons d’une portée avec une autre
Chez le chien le sexe sur le certificat est déterminé par le marqueur AMELOGENIN.
| Sexe | STR, en lettre | STR, en chiffre | SNP |
|---|---|---|---|
| Mâle | X/Y ou Y/X | 182/217 | A/G |
| Femelle | X/X | 217/217 | G/G |
Le sexe transmis à la déclaration n’est pas pris en compte. L’ADN présent dans le tube d’une part et le numéro de puce (ou autre identification) sur le même tube d’autre part sont inextricablement liés. Lorsqu’il s’écarte du vrai sexe, un échange lors du prélèvement ou de la réception des prélèvements est à exclure.
Et maintenant?
Tout d’abord, les données d’analyse brutes seront revues pour voir si les signaux ont bien été interprétés. Le cas échéant, bien sûr, il sera corrigé sans frais supplémentaires.
Dans le cas d’un échange non seulement le sexe est faux mais le profil entier. Le mauvais animal était après tout lié au mauvais tube.
Nous vérifions l’identification des tubes d’origine et les échantillons sont analysés à nouveau. Après il reste deux possibilités:
-
- La réanalyse entraîne un autre profil (et sexe). L’erreur s’est produite avec la réception des échantillons. Elle est corrigée sans frais supplémentaires.
- La réanalyse entraîne un même profil (et sexe). L’erreur s’est produite pendant le prélèvement. Elle ne peut pas être corrigée sans un nouveau prélèvement à ses frais.
Pour des raisons de confidentialité et de déontologie, les rapports d’analyse sont uniquement envoyés à:
- le vétérinaire demandeur ou l’expéditeur des échantillons
- le propriétaire de l’animal au moment de la demande
- toutes adresses e-mail en cc au moment de la demande
Une tierce partie non impliquée dans la demande initiale ne peut pas recevoir une copie en direct, mais seulement par l’intermédiaire du vétérinaire ou du propriétaire d’origine.
En tout cas nous conserveront le droit de demander le paiement préalable des analyses effectuées ou à effectuer.

 p.ex. pedigree concluant
p.ex. pedigree concluant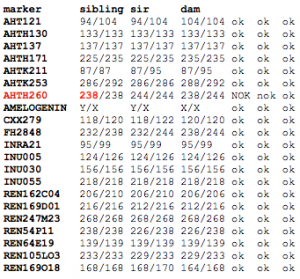 p.ex. un décalage par rapport au père
p.ex. un décalage par rapport au père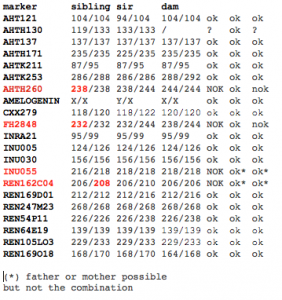 p.ex. 4 marqueurs échouent par rapport à la mère
p.ex. 4 marqueurs échouent par rapport à la mère